Une nouvelle méthode découvre un biomarqueur de la maladie inflammatoire de l’intestin
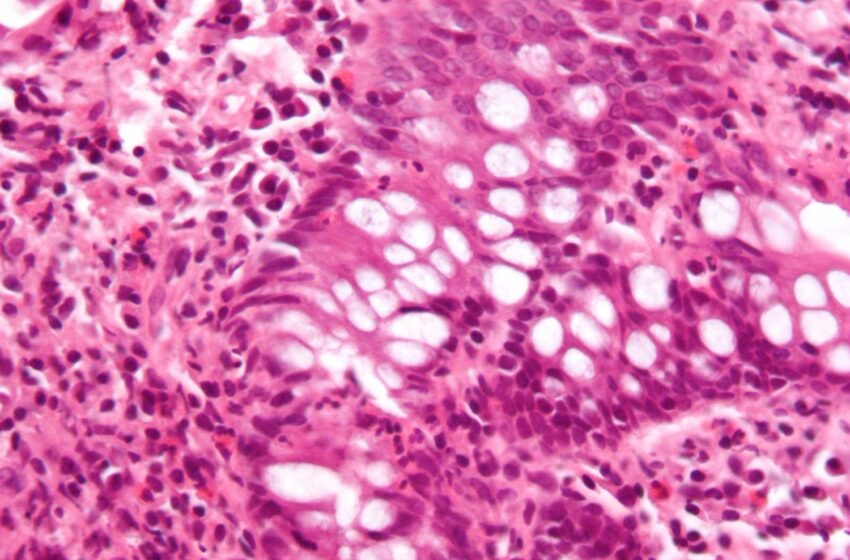
Micrographie montrant une inflammation du gros intestin dans un cas de maladie inflammatoire de l'intestin. Biopsie colique. Crédit : Wikipédia/CC BY-SA 3.0
Ces dernières années, la recherche sur le microbiome a commencé à se déplacer des microbes eux-mêmes vers les molécules qu’ils produisent. Après tout, ce sont ces molécules qui interagissent directement avec les cellules humaines pour influencer la santé d'une personne. Cependant, tenter d’identifier quelles molécules sont fabriquées par le microbiome d’une personne est un véritable défi. Une étude métabolomique typique ne peut caractériser qu’environ 10 % des données moléculaires d’un échantillon de microbiome humain.
Dans une nouvelle étude publiée dans Nature, des experts en microbiome de l'Université de Californie à San Diego lancent une nouvelle approche qu'ils appellent « métabolomique inverse ». La technique combine la synthèse organique, la science des données et la spectrométrie de masse pour mieux comprendre quelles molécules sont sécrétées par le microbiome et comment elles affectent la santé humaine.
Dans leur première application de la métabolomique inverse, les scientifiques ont découvert des centaines de molécules qui n’avaient jamais été observées auparavant dans le corps humain. Grâce à ces nouvelles données, ils ont pu identifier une nouvelle signature métabolomique pour les maladies inflammatoires de l’intestin (MII). Les auteurs affirment que ces molécules pourraient un jour servir de biomarqueur pour diagnostiquer les MII ou de cible thérapeutique potentielle pour aider à traiter la maladie.
“Nous savons que le microbiome est important, mais nous ne savons pas quels types de molécules les microbes produisent ni quelle influence ils ont sur le corps humain”, a déclaré l'auteur principal Pieter C. Dorrestein, Ph.D., professeur à la Skaggs School of Pharmacie et sciences pharmaceutiques à l'UC San Diego. “La métabolomique inverse nous aide à évaluer si des molécules spécifiques peuvent être trouvées dans les échantillons, à prédire quels microbes les produisent et à relier ces signatures métabolomiques à la santé et à la maladie.”
Dans une étude métabolomique typique, les chercheurs utiliseront un outil appelé spectrométrie de masse pour rechercher différentes molécules dans un échantillon. Dans cette technique, chaque molécule possède son propre « code-barres » grâce auquel elle peut être identifiée. Néanmoins, les scientifiques ont besoin de savoir ce que représentent ces codes-barres pour décrire le contenu d’un échantillon, ce qui reste un défi.
Dans la nouvelle étude, les chercheurs du Dorrestein Lab ont adopté une approche rétrospective. La première auteure Emily C. Gentry, Ph.D., aujourd'hui professeure adjointe à Virginia Tech, a utilisé la synthèse organique pour produire d'abord des milliers de molécules synthétiques différentes provenant de quatre classes d'intérêt, puis a défini chacun de leurs codes-barres.
Les chercheurs ont ensuite utilisé des données métabolomiques accessibles au public, y compris celles précédemment collectées par la Crohn's & Colitis Foundation, et ont recherché les nouveaux codes-barres dans ces données. Les résultats ont révélé que 145 des acides biliaires synthétisés étaient présents dans des échantillons biologiques provenant de données publiques, dont 139 n'avaient jamais été décrits auparavant.
“Si vous lisez un manuel de biologie, aucune de ces molécules n'y figurera”, a déclaré Dorrestein. “Non seulement ils sont nouveaux dans notre compréhension de la physiologie humaine, mais ils sont également entièrement nouveaux pour la science, ce qui est assez étonnant.”
Gentry et ses collègues ont ensuite comparé les signatures métabolomiques d'échantillons provenant de différentes populations de patients et ont découvert une forte association entre une classe synthétisée de molécules microbiennes – les amidates biliaires – et les MII. Cette association a ensuite été validée sur plusieurs cohortes, confortant l’idée selon laquelle ces molécules sont probablement impliquées dans la pathologie des MII.
En y regardant de plus près, les scientifiques ont remarqué que certains amidates biliaires étaient élevés chez les patients atteints de la maladie de Crohn, en particulier lorsqu'ils présentaient des symptômes actifs, mais ce n'était pas le cas pour les patients atteints de colite ulcéreuse. Des modèles comme ceux-ci pourraient un jour être utilisés pour aider à différencier et à diagnostiquer des types spécifiques de MII.
Les chercheurs ont ensuite commencé à explorer comment ces molécules pourraient influencer la santé intestinale. Des expériences supplémentaires ont montré que plusieurs composés d’amidate biliaire peuvent favoriser l’inflammation intestinale en dérégulant la fonction des lymphocytes T. Par exemple, un composé microbien a produit une multiplication par six d'une cytokine clé connue pour être impliquée dans la pathogenèse de la maladie de Crohn.
“Nous utilisons la synthèse organique et la science des données pour mieux comprendre le fonctionnement de notre corps au niveau moléculaire”, a déclaré Gentry. “Nous sommes également l'une des premières études à découvrir de nouvelles molécules humaines à l'aide de données métabolomique accessibles au public. À mesure que davantage de données métabolomique seront accessibles au public, la métabolomique inverse deviendra encore plus informative.”
Les auteurs affirment que les molécules qu’ils ont décrites pourraient un jour inspirer de nouveaux traitements pour le traitement des MII. Par exemple, les patients peuvent être traités avec des pilules contenant des microbes vivants qui sécrètent des molécules spécifiques ou des médicaments qui inhibent les enzymes avec lesquelles ces molécules associées à la maladie interagissent.
« Il s'agit d'une réalisation remarquable issue de notre initiative de nutrition de précision, dans laquelle le Dr Dorrestein a précédemment démontré que la métabolomique inverse pouvait identifier les métabolites alimentaires associés à la gravité de la maladie chez les patients atteints de MII », a déclaré Andrés Hurtado-Lorenzo, Ph.D., vice-président président de Translational Research & IBD Ventures à la Crohn's & Colitis Foundation. « Maintenant, ces travaux révolutionnaires ont progressé vers la découverte de nouveaux métabolites qui présentent un potentiel pour des applications diagnostiques et thérapeutiques dans les MII. »
Plus d'information:
Emily C. Gentry et al, Métabolomique inverse pour la découverte de structures chimiques chez l'homme, Nature (2023). DOI : 10.1038/s41586-023-06906-8
Fourni par l'Université de Californie – San Diego
Citation: Métabolomique inverse : une nouvelle méthode trouve un biomarqueur pour la maladie inflammatoire de l'intestin (5 décembre 2023) récupéré le 5 décembre 2023 sur
Ce document est soumis au droit d'auteur. En dehors de toute utilisation équitable à des fins d'étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.
