Une étude montre que les cellules T peuvent lutter contre la nouvelle variante du SRAS-CoV-2 « Pirola »
Résumé graphique. Crédit: Hôte cellulaire et microbe (2023). DOI : 10.1016/j.chom.2023.11.010
En août, des chercheurs ont détecté une nouvelle « variante préoccupante » du SRAS-CoV-2 chez des patients en Israël et au Danemark. Depuis, cette variante, baptisée BA.2.86 ou « Pirola », a fait le tour du monde. Le variant Pirola a sonné l’alarme car il est fortement muté. En fait, Pirola est aussi muté que la variante omicron, comparée à la première variante du SRAS-CoV-2 incluse dans les vaccinations originales.
Alors que Pirola se propage, les chercheurs de l’Institut d’immunologie de La Jolla (LJI) étudient si les vaccins contre le COVID-19 (ou une exposition antérieure au SRAS-CoV-2) peuvent encore protéger les personnes contre une maladie grave.
“On craint qu'un virus présentant un nombre aussi élevé de mutations” échappe “à l'immunité des lymphocytes T”, déclare le professeur Alessandro Sette, Dr. Biol.Sci, du LJI.
Maintenant, une nouvelle étude dans Hôte cellulaire et microbe suggère que les cellules T peuvent voir à travers les mutations de Pirola et trouver leurs cibles. “Notre analyse suggère qu'il y a des nouvelles positives”, déclare Alba Grifoni, Ph.D., professeur adjoint de recherche au LJI. “Il semble qu'une exposition antérieure à l'omicron – ou une vaccination avec les vaccins bivalents les plus récents – puisse armer une personne avec des lymphocytes T capables de” rattraper “et de générer des réponses spécifiques pour combattre Pirola.”
Exploiter la bioinformatique
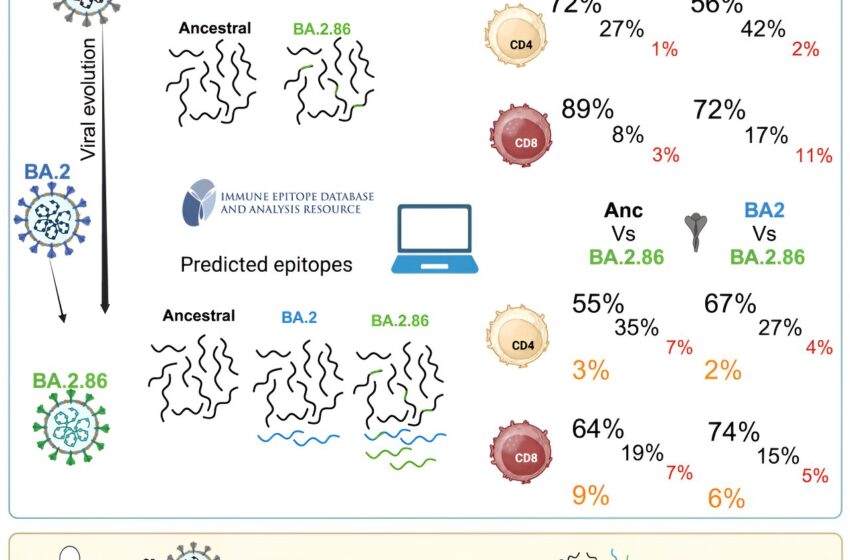
Pour la nouvelle étude, Sette et Grifoni se sont tournés vers une ressource appelée Immune Epitope Database (IEDB). Cette base de données contient des découvertes précieuses, recueillies par des immunologistes du monde entier, décrivant comment les cellules immunitaires reconnaissent des fragments, ou « épitopes », sur les microbes.
Grâce à la richesse des données de l'IEDB, les chercheurs disposaient déjà d'une image détaillée de la manière dont les vaccins contre le COVID-19 ou une exposition antérieure au SRAS-CoV-2 « entraînaient » les cellules T à cibler les épitopes du SRAS-COV-2. Les chercheurs ont extrait ces données IEDB et développé un pipeline bioinformatique pour prédire comment ces cellules T réagiraient à la variante Pirola.
“Nous avons simulé la réponse des lymphocytes T au Pirola sur la base de données expérimentales et prédites des variantes précédentes du SRAS-CoV-2”, explique Grifoni.
Les chercheurs ont découvert que la plupart des lymphocytes T pouvaient encore cibler les épitopes de Pirola :
- Dans l'ensemble, 72 pour cent des fragments reconnus par CD4+ Réponses des lymphocytes T « auxiliaires » et 89 pour cent des CD8+ Les épitopes des lymphocytes T « tueurs » étaient inchangés, ou « conservés », entre les variantes.
- Les chercheurs ont trouvé moins d’épitopes de lymphocytes T conservés sur la protéine « Spike » de Pirola, comme prévu étant donné qu’elle héberge la plupart des mutations. Seulement 56 pour cent des CD4+ Epitopes de lymphocytes T « auxiliaires » et CD8+ Les épitopes des lymphocytes T « tueurs » ont été conservés sur cette protéine structurelle majeure. Il s’agissait d’un problème potentiel, car les vaccins actuels contre le COVID-19 sont conçus uniquement pour apprendre aux cellules immunitaires à reconnaître et à cibler les épitopes Spike.
- Pourtant, lorsque les chercheurs ont examiné de plus près les fragments Spike, ils ont constaté que 96 pour cent des CD4+ Epitopes de lymphocytes T « auxiliaires » et 62 % des CD8+ Les épitopes des lymphocytes T « tueurs » étaient suffisamment similaires pour que les lymphocytes T puissent probablement encore les reconnaître.
Bref, si Pirola veut échapper aux cellules T, il ne fait pas un très bon travail.
“Un grand nombre d'épitopes reconnus par le système immunitaire sont encore conservés sur la nouvelle variante Pirola”, explique Sette. “Nous prévoyons fortement que le virus sera toujours reconnu par les cellules T.”
“Les lymphocytes T pourraient également être capables de “courir” après les peptides nouvellement mutés de Pirola pour développer une nouvelle réponse contre ces épitopes, comme nous l'avons vu pour d'autres variantes”, ajoute Grifoni. “Nous pensons que cela peut expliquer pourquoi, malgré l'évolution virale, nous n'avons pas vu de maladie plus grave dans les cas d'infection à Pirola ou d'autres variantes plus récentes.”
Prochaines étapes
Grifoni souligne que ces résultats sont des prédictions et non des observations basées sur des infections réelles à Pirola. Elle pense néanmoins qu’il est important de voir comment ces prédictions « in silico » (dans un ordinateur) se reflètent dans les études récentes du monde réel. “Nous avons encore besoin d'une validation expérimentale, mais nous avons établi plusieurs collaborations dans le monde entier qui étudient cette question à l'heure où nous parlons”, explique Grifoni.
De nombreuses personnes sont encore vulnérables à l’infection par le SRAS-COV-2, même par le variant Pirola, ajoute Sette. “C'est pourquoi les gens devraient continuer à être vaccinés, en particulier avec les vaccins mis à jour.”
Les chercheurs collectent actuellement des données expérimentales pour en savoir plus sur les réponses des lymphocytes T aux variantes et renforcer davantage leurs outils de prédiction. Grifoni est particulièrement curieux de comprendre exactement comment les personnes qui ont reçu des rappels de vaccin bivalent et/ou des infections « révolutionnaires » développent des réponses à lymphocytes T contre de futures variantes.
Plus d'information:
Alessandro Sette et al, Il est prévu que les cellules T spécifiques du SRAS-2 préexistantes reconnaissent BA.2.86, Hôte cellulaire et microbe (2023). DOI : 10.1016/j.chom.2023.11.010. www.cell.com/cell-host-microbe… 1931-3128(23)00460-2
Fourni par l'Institut d'immunologie de La Jolla
Citation: Une étude montre que les cellules T peuvent lutter contre la nouvelle variante « Pirola » du SRAS-CoV-2 (8 décembre 2023) récupérée le 8 décembre 2023 sur
Ce document est soumis au droit d'auteur. En dehors de toute utilisation équitable à des fins d'étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.
