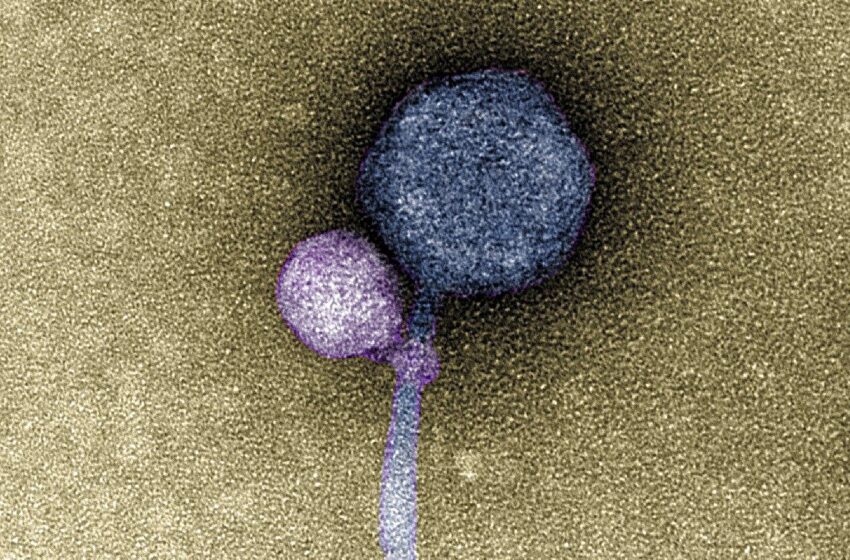
Première observation d’un virus s’attachant à un autre virus
Cette image colorisée au microscope électronique à transmission montre un virus satellite récemment découvert accroché à son virus auxiliaire. Cette recherche représente la première fois que des scientifiques observent un virus attaché à un autre. Sur 50 assistants observés, 40 avaient un satellite. L’analyse bioinformatique des génomes des virus satellite et assistant fournit des indices sur la raison pour laquelle le satellite a pu évoluer pour s’attacher au virus assistant, et suggère que cette paire pourrait avoir co-évolué pendant environ 100 millions d’années. Crédit : Tagide deCarvalho
Personne n’avait jamais vu un virus s’accrocher à un autre virus, jusqu’à ce que des résultats de séquençage anormaux envoient une équipe de l’UMBC dans un terrier de lapin, menant à une découverte unique en son genre.
On sait que certains virus, appelés satellites, dépendent non seulement de leur organisme hôte pour compléter leur cycle de vie, mais aussi d’un autre virus, appelé “assistant”, explique Ivan Erill, professeur de sciences biologiques. Le virus satellite a besoin d’aide soit pour construire sa capside, une coque protectrice qui renferme le matériel génétique du virus, soit pour l’aider à répliquer son ADN.
Ces relations virales nécessitent que le satellite et l’assistant soient proches l’un de l’autre au moins temporairement, mais il n’y avait aucun cas connu où un satellite s’est réellement attaché à un assistant – jusqu’à présent.
Dans un article publié dans La revue ISMEune équipe de l’UMBC et des collègues de l’Université de Washington à St. Louis (WashU) décrivent la première observation d’un bactériophage satellite (un virus qui infecte les cellules bactériennes) s’attachant systématiquement à un bactériophage auxiliaire au niveau de son « cou », là où la capside rejoint la queue. du virus.
Dans les images détaillées de microscopie électronique prises par Tagide deCarvalho, directeur adjoint des installations principales du Collège des sciences naturelles et mathématiques et premier auteur du nouvel article, 80 pour cent (40 sur 50) des assistants avaient un satellite attaché au cou. Certains de ceux qui n’en avaient pas avaient des vrilles satellites restantes présentes au niveau du cou. Erill, auteur principal de l’article, les décrit comme ressemblant à des « marques de morsure ».
“Quand je l’ai vu, je me suis dit, je ne peux pas y croire”, dit deCarvalho. “Personne n’a jamais vu un bactériophage, ni aucun autre virus, s’attacher à un autre virus.”
Une relation virale à long terme
Après les premières observations, Elia Mascolo, étudiant diplômé du groupe de recherche d’Erill et co-premier auteur de l’article, a analysé les génomes du satellite, de l’assistant et de l’hôte, ce qui a révélé d’autres indices sur ce virus inédit. relation. La plupart des virus satellites contiennent un gène qui leur permet de s’intégrer dans le matériel génétique de la cellule hôte après y avoir pénétré. Cela permet au satellite de se reproduire chaque fois qu’un assistant entre dans la cellule. La cellule hôte copie également l’ADN du satellite avec le sien lorsqu’elle se divise.
Un échantillon de bactériophage de WashU contenait également un assistant et un satellite. Le satellite WashU possède un gène d’intégration et ne s’attache pas directement à son assistant, à l’instar des systèmes satellite-assistant observés précédemment.
Cependant, le satellite de l’échantillon de l’UMBC, nommé MiniFlayer par les étudiants qui l’ont isolé, est le premier cas connu de satellite sans gène d’intégration. Parce qu’il ne peut pas s’intégrer dans l’ADN de la cellule hôte, il doit se trouver à proximité de son assistant, nommé MindFlayer, chaque fois qu’il pénètre dans une cellule hôte pour survivre. Étant donné que, même si l’équipe n’a pas directement prouvé cette explication, “l’attachement était désormais tout à fait logique”, dit Erill, “car sinon, comment allez-vous garantir que vous entrerez dans la cellule en même temps ?”
Analyse bioinformatique supplémentaire par Mascolo et Julia López-Pérez, une autre doctorante. étudiant travaillant avec Erill, a révélé que MindFlayer et MiniFlayer coévoluent depuis longtemps. “Ce satellite écoute et optimise son génome pour qu’il soit associé à l’assistant depuis, je dirais, au moins 100 millions d’années”, explique Erill, ce qui suggère qu’il pourrait y avoir de nombreux autres cas de ce type de relation à découvrir. .
Contamination ou découverte ?
Cette découverte révolutionnaire aurait facilement pu passer inaperçue. Le projet a débuté comme un semestre typique du programme SEA-PHAGES, un programme d’investigation dans lequel les étudiants de premier cycle isolent des bactériophages à partir d’échantillons environnementaux, les envoient pour séquençage, puis utilisent des outils bioinformatiques pour analyser les résultats. Lorsque le laboratoire de séquençage de l’Université de Pittsburgh a signalé une contamination dans l’échantillon de l’UMBC censé contenir le phage MindFlayer, le voyage a commencé.
L’échantillon comprenait une grande séquence : le phage attendu. “Mais au lieu de simplement découvrir cela, nous avons également trouvé une petite séquence, qui ne correspondait à rien de ce que nous connaissions”, explique Erill, qui est également l’un des responsables du programme SEA-PHAGES de l’UMBC, appelé Phage Hunters, avec Steven Caruso, maître de conférences en sciences biologiques. Caruso a recommencé l’isolement, l’a envoyé pour séquençage et a obtenu des résultats identiques.
C’est à ce moment-là que l’équipe a fait appel à deCarvalho pour obtenir un aperçu de ce qui se passait avec le microscope électronique à transmission (TEM) du centre d’imagerie Keith R. Porter (KPIF) de l’UMBC. Sans les images, la découverte aurait été impossible.
“Tout le monde ne dispose pas d’un TEM”, note deCarvalho. Mais grâce aux instruments du KPIF, deCarvalho déclare : “Je suis capable de suivre certaines de ces observations et de les valider par imagerie. Il y a des éléments de découverte que nous ne pouvons réaliser qu’en utilisant le TEM.”
La découverte de l’équipe ouvre la voie à de futurs travaux visant à comprendre comment le satellite s’attache, à quel point ce phénomène est courant et bien plus encore. “Il est possible qu’un grand nombre de bactériophages que les gens pensaient contaminés soient en réalité ces systèmes d’assistance par satellite”, explique deCarvalho, “donc maintenant, avec cet article, les gens pourraient être en mesure de reconnaître davantage de ces systèmes.”
Plus d’information:
Tagide deCarvalho et al, Entrée simultanée comme adaptation à la virulence dans un nouveau système d’assistance par satellite infectant les espèces de Streptomyces, La revue ISME (2023). DOI : 10.1038/s41396-023-01548-0
Fourni par l’Université du Maryland Comté de Baltimore
Citation: Première observation d’un virus s’attachant à un autre virus (2 novembre 2023) récupéré le 2 novembre 2023 sur
Ce document est soumis au droit d’auteur. En dehors de toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.
