Une nouvelle approche de l’IA pour la conception des protéines
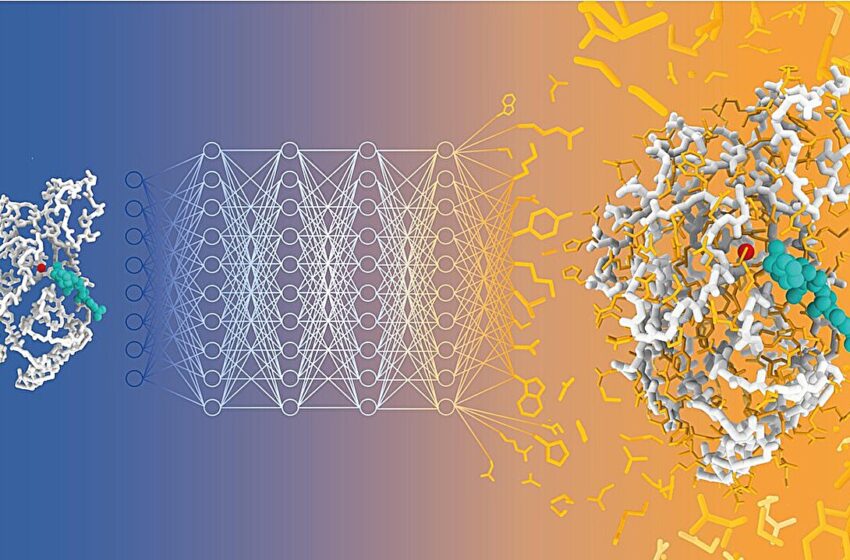
Représentation schématique de la prédiction de séquence avec CARBonAra. Le transformateur géométrique échantillonne l’espace de séquence de l’enzyme bêta-lactamase TEM-1 (en gris) complexée à un substrat naturel (en cyan) pour produire de nouvelles enzymes bien repliées et actives. Crédit : Alexandra Banbanaste (EPFL)
Des chercheurs de l’EPFL ont développé un nouveau modèle basé sur l’intelligence artificielle, conçu pour prédire les séquences protéiques à partir de structures structurales, en intégrant des environnements moléculaires complexes. Ce modèle promet des avancées significatives dans l’ingénierie des protéines et des applications dans divers domaines, notamment la médecine et la biotechnologie.
Concevoir des protéines capables d’accomplir des fonctions spécifiques implique de comprendre et de manipuler leurs séquences et leurs structures. Cette tâche est essentielle pour développer des traitements ciblés contre les maladies et créer des enzymes pour des applications industrielles.
L’un des grands défis de l’ingénierie des protéines est de concevoir des protéines de novo, c’est-à-dire de toutes pièces, afin d’adapter leurs propriétés à des tâches spécifiques. Cela a de profondes implications pour la biologie, la médecine et la science des matériaux. Par exemple, les protéines modifiées peuvent cibler des maladies avec une grande précision, offrant ainsi une alternative compétitive aux médicaments traditionnels à base de petites molécules.
De plus, des enzymes conçues sur mesure, qui agissent comme des catalyseurs naturels, peuvent faciliter des réactions rares ou inexistantes dans la nature. Cette capacité est particulièrement précieuse dans l’industrie pharmaceutique pour synthétiser des molécules médicamenteuses complexes et dans les technologies environnementales pour décomposer plus efficacement les polluants ou les plastiques.
Une équipe de scientifiques dirigée par Matteo Dal Peraro de l’EPFL a développé CARBonAra (Context-aware Amino acid Recovery from Backbone Atoms and heteroatoms), un modèle piloté par l’IA qui peut prédire les séquences de protéines, mais en tenant compte des contraintes imposées par différents environnements moléculaires – une réalisation unique.
CARBonAra est formé sur un ensemble de données d’environ 370 000 sous-unités, avec 100 000 supplémentaires pour la validation et 70 000 pour les tests provenant de la Protein Data Bank (PDB). La recherche est publiée dans la revue Nature Communications.
CARBonAra s’appuie sur l’architecture du framework Protein Structure Transformer (PeSTo), également développé par Lucien Krapp dans le groupe de Dal Peraro. Il utilise des transformateurs géométriques, qui sont des modèles d’apprentissage profond qui traitent les relations spatiales entre les points, comme les coordonnées atomiques, pour apprendre et prédire des structures complexes.
CARBonAra peut prédire les séquences d’acides aminés à partir des structures de base, les cadres structuraux des molécules protéiques. Cependant, l’une des caractéristiques les plus remarquables de CARBonAra est sa prise en compte du contexte, qui se manifeste notamment par la façon dont il améliore les taux de récupération des séquences, c’est-à-dire le pourcentage d’acides aminés corrects prédits à chaque position d’une séquence protéique par rapport à une séquence de référence connue.
CARBonAra améliore considérablement les taux de récupération lorsqu’il inclut des « contextes » moléculaires, tels que les interfaces protéiques avec d’autres protéines, des acides nucléiques, des lipides ou des ions. « Cela est dû au fait que le modèle est entraîné avec toutes sortes de molécules et s’appuie uniquement sur des coordonnées atomiques, de sorte qu’il peut gérer non seulement les protéines », explique Dal Peraro. Cette fonctionnalité améliore à son tour la puissance prédictive du modèle et son applicabilité dans des systèmes biologiques complexes et réels.
Le modèle ne se révèle pas seulement performant dans les tests synthétiques, mais il a été validé expérimentalement. Les chercheurs ont utilisé CARBonAra pour concevoir de nouvelles variantes de l’enzyme β-lactamase TEM-1, impliquée dans le développement de la résistance aux antimicrobiens.
Certaines des séquences prédites, différant d’environ 50 % de la séquence de type sauvage, ont été repliées correctement et conservent une certaine activité catalytique à haute température, lorsque l’enzyme de type sauvage est déjà inactive.
La flexibilité et la précision de CARBonAra ouvrent de nouvelles perspectives pour l’ingénierie des protéines. Sa capacité à prendre en compte des environnements moléculaires complexes en fait un outil précieux pour la conception de protéines aux fonctions spécifiques, améliorant ainsi les futures campagnes de découverte de médicaments. De plus, le succès de CARBonAra dans l’ingénierie enzymatique démontre son potentiel pour les applications industrielles et la recherche scientifique.
Plus d’information:
Lucien F. Krapp et al, Apprentissage profond géométrique sensible au contexte pour la conception de séquences protéiques, Nature Communications (2024). DOI: 10.1038/s41467-024-50571-y
Fourni par l’Ecole Polytechnique Fédérale de Lausanne
Citation:Une nouvelle approche de l’IA pour la conception des protéines (2024, 7 août) récupéré le 7 août 2024 à partir de
Ce document est soumis au droit d’auteur. En dehors de toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni à titre d’information uniquement.
