Des modèles informatiques sophistiqués ouvrent la porte à des antibiotiques bien plus ciblés
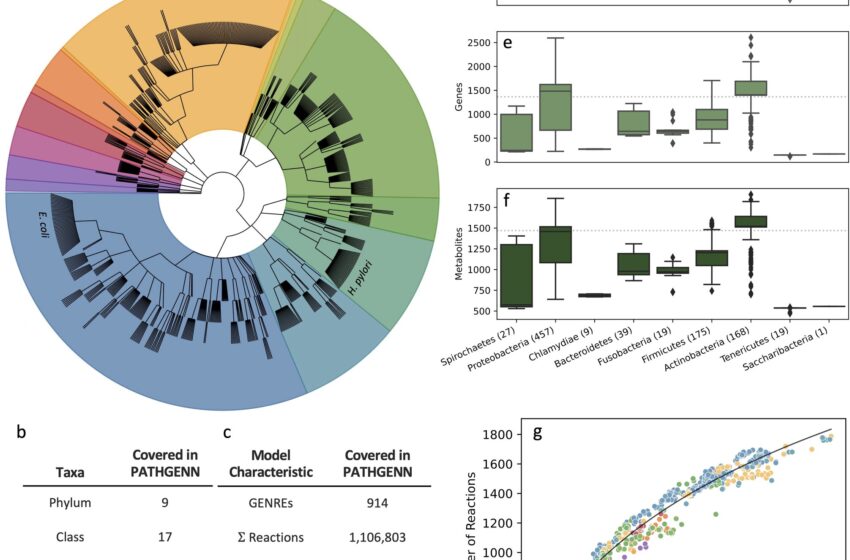
Portée de notre collection de modèles pathogènes du métabolisme. Crédit: Biologie PLOS (2024). DOI : 10.1371/journal.pbio.3002907
La résistance aux antibiotiques étant un problème croissant, les chercheurs de la Faculté de médecine de l’Université de Virginie ont développé des modèles informatiques de pointe qui pourraient donner aux médicaments anti-maladie une précision comparable à celle d’un laser pour cibler uniquement des bactéries spécifiques dans des parties spécifiques du corps.
Dans l’état actuel des choses, les antibiotiques tuent les bactéries sans discernement. Parce que ces médicaments sont largement utilisés, un nombre croissant d’insectes dangereux deviennent résistants, menaçant l’une des armes les plus importantes de la médecine moderne contre la maladie.
La nouvelle approche d’UVA, en revanche, limiterait considérablement la fréquence à laquelle les bactéries sont exposées aux antibiotiques, réduisant ainsi le risque qu’elles deviennent résistantes aux antibiotiques. De plus, cette approche représenterait un pas en avant significatif pour la médecine de précision, permettant aux médecins de mieux adapter les traitements aux besoins de chaque patient. Au lieu de prendre un antibiotique qui tue les bactéries, qu’elles soient utiles ou nocives, les patients pourraient recevoir des antibiotiques ciblant des bactéries spécifiques causant un problème spécifique dans une zone spécifique du corps.
“De nombreux défis biomédicaux sont incroyablement complexes et les modèles informatiques apparaissent comme un outil puissant pour résoudre ces problèmes”, a déclaré le chercheur Jason Papin, Ph.D., du département de génie biomédical de l’UVA. “Nous espérons que ces modèles informatiques des réseaux moléculaires des bactéries nous aideront à développer de nouvelles stratégies pour traiter les infections.”
Les chercheurs ont publié leurs résultats dans la revue Biologie PLOS.
Des antibiotiques plus ciblés
La nouvelle approche d’UVA a été rendue possible grâce aux efforts herculéens de Papin, Ph.D. l’étudiante Emma Glass et leurs collaborateurs. En collaboration avec Andrew Warren, Ph.D., du Biocomplexity Institute de l’UVA, les chercheurs du laboratoire de Papin ont développé des modèles informatiques sophistiqués de chaque pathogène bactérien humain avec suffisamment d’informations génétiques disponibles.
Glass a ensuite analysé tous ces modèles et identifié des traits communs parmi les bactéries. Cette analyse a permis de découvrir que les bactéries présentes dans certaines parties du corps, comme l’estomac, avaient tendance à partager des propriétés métaboliques. Fondamentalement, l’endroit où ils vivent détermine leur fonctionnement.
“En utilisant nos modèles informatiques, nous avons découvert que les bactéries vivant dans l’estomac avaient des propriétés uniques”, a déclaré Glass. “Ces propriétés peuvent être utilisées pour guider la conception d’antibiotiques ciblés, qui pourraient un jour, espérons-le, ralentir l’émergence d’infections résistantes.”
Les similitudes communes entre les microbes de différents endroits pourraient être le talon d’Achille des bactéries nocives dans notre corps. Grâce à des recherches plus approfondies, les médecins pourraient être en mesure de cibler des types spécifiques de bactéries dans des zones spécifiques, réduisant ainsi le besoin d’antibiotiques à large spectre.
En mettant à l’épreuve leur approche de modélisation informatique, Papin et son équipe ont déjà découvert qu’ils pouvaient inhiber la croissance de punaises d’estomac nuisibles lors d’expériences en laboratoire. C’est un signe prometteur pour le potentiel futur de leur approche de modélisation informatique.
“Nous avons encore beaucoup à faire pour tester ces idées sur d’autres bactéries et types d’infections”, a déclaré Papin. “Mais ce travail montre l’incroyable promesse de la science des données et de la modélisation informatique pour résoudre certains des problèmes les plus importants de la recherche biomédicale.”
L’équipe de recherche était composée de Glass, Lillian R. Dillard, Glynis L. Kolling, Warren et Papin. Les scientifiques n’ont aucun intérêt financier dans ces travaux.
Plus d’informations :
Emma M. Glass et al, Les phénotypes métaboliques spécifiques à une niche peuvent être utilisés pour identifier des cibles antimicrobiennes chez les agents pathogènes, Biologie PLOS (2024). DOI : 10.1371/journal.pbio.3002907
Fourni par l’Université de Virginie
Citation: Des modèles informatiques sophistiqués ouvrent la porte à des antibiotiques beaucoup plus ciblés (14 janvier 2025) récupéré le 14 janvier 2025 sur
Ce document est soumis au droit d’auteur. En dehors de toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni à titre informatif uniquement.
