Les chercheurs déterminent la base structurelle des interactions ApoE-Aβ
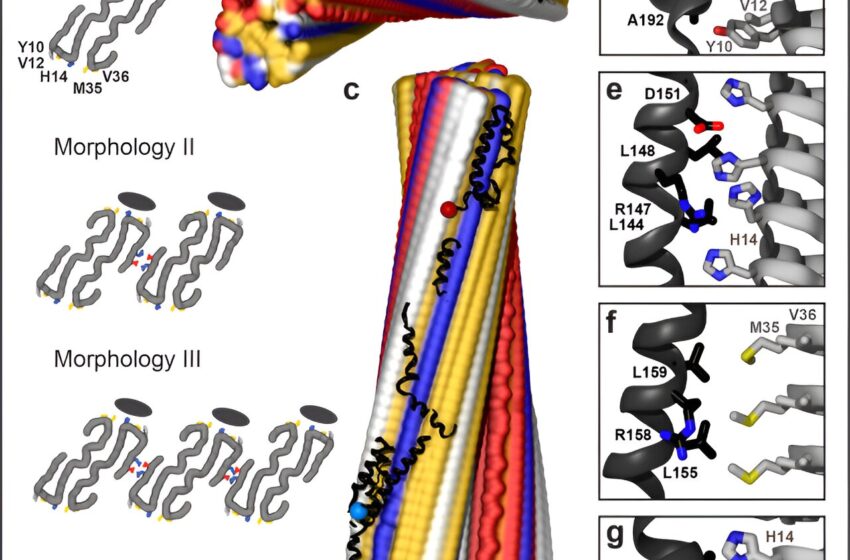
Modèle structurel de l'apoE ancré sur Aβ1 à 40 fibrille du système vasculaire AD. Le modèle a été obtenu en utilisant les segments apoE présentés dans les figures supplémentaires 2d – h et la structure des fibrilles (ID PDB : 6SHS). Les positions des segments sont compatibles avec l’apoE complète. (un) Morphologies de fibrilles I-III contenant un à trois protofilaments appariés (chaîne principale en gris, vue vers le bas de l'axe des fibrilles). Morphologie J'ai été utilisé pour l'accostage. Les ovales noirs marquent la position d'amarrage de l'apoE sur l'un des deux sites prévus par protofilament apparié. Résidus de coordination de l'ApoE Y10, V12, H14 de la molécule 1 et M35, V36 de la molécule 2 de Aβ1 à 40 les protofilaments sont indiqués. Dans les morphologies II et III, les paires de résidus E3 et R5 qui forment des ponts salins entre protofilaments adjacents (63) sont présentées. (avant JC) Les vues de dessus et de côté du modèle d'amarrage montrent apoE aux côtés de Aβ1 à 40 protofilament. Dans cette figure et dans d’autres figures de complexes apolipoprotéine-amyloïde, les chaînes principales d’apolipoprotéine sont en rubans noirs ; des points bleus et rouges marquent les terminaisons N et C. Les fibrilles amyloïdes sont représentées en surface : jaune – hydrophobe, blanche – polaire, rouge – acide, bleue – basique (y compris His). Les panneaux dg montrent les contacts apoE-amyloïde dans environ 5 Å dans les régions sélectionnées (apoE – noir, Aβ – gris). (d) Aβ1 à 40 l'échelle de résidus Y10, V12 forme des interactions hydrophobes avec les résidus apoE ; A192, V195, L198, A199 de la région charnière apoE sont illustrés. (e) H14 de Aβ1 à 40 forme des interactions mixtes (polaires/hydrophobes) avec les résidus apoE ; R147, L144, L148, D151 de l'hélice 4 sont représentés. (F) R158 dans l'apoE3/E4, qui flanque la face hydrophobe de l'hélice 4, interagit défavorablement avec M35 de Aβ1 à 40. (g) Les résidus hydrophobes de l'hélice 3 de l'apoE interagissent avec les échelles de résidus de V12 et H14 dans Aβ1 à 40. Le résidu 112 (R112 dans l’apoE4) s’éloigne de l’amyloïde. Crédit: Sciences de la vie cellulaire et moléculaire (2023). DOI : 10.1007/s00018-023-05026-w
La maladie d'Alzheimer (MA) est une maladie incurable et débilitante qui touche des millions de patients dans le monde. Les dépôts d’amyloïde bêta (Aβ) dans le cerveau sont la clé de la pathologie de la MA à un stade précoce. L'apolipoprotéine E (apoE) interagit avec l'Aβ et peut influencer ce processus pathologique. Bien que les interactions apoE-Aβ aient été étudiées de manière approfondie et proposées comme cible thérapeutique dans la MA, les résultats des études antérieures prêtent à confusion. Certains rapportent que l’apoE favorise la formation d’amyloïde Aβ tandis que d’autres signalent que l’apoE bloque ce processus pathologique.
Une nouvelle étude biophysique menée par des chercheurs de la Chobanian & Avedisian School of Medicine de l'Université de Boston explique pourquoi l'apoE et d'autres apolipoprotéines co-déposent avec les amyloïdes et fournit une base structurelle pour comprendre comment les apolipoprotéines modulent la croissance et la prolifération amyloïde dans le corps.
“Notre analyse de quatre fibrilles amyloïdes différentes montre que les interactions apoE-amyloïde dépendent de la structure amyloïde exacte, appelée polymorphe. Les polymorphes amyloïdes sont spécifiques à la maladie et aux tissus et peuvent différer selon les expériences, ce qui explique les divergences entre les études antérieures. Comprendre une telle maladie -des facteurs spécifiques sont essentiels pour exploiter les interactions apoE-Aβ à des fins de ciblage thérapeutique”, explique la première auteure Emily Lewkowicz, Ph.D. candidat en biophysique à l'école.
Les chercheurs ont utilisé quatre structures différentes de fibrilles Aβ provenant du cerveau de patients atteints de la maladie d'Alzheimer et d'autres maladies neurodégénératives. Pour construire des modèles d'apoE dans des complexes avec différentes fibrilles, ils ont effectué un docking protéique (la prédiction de la structure du complexe basée sur les structures des protéines individuelles) à l'aide d'une modélisation informatique, suivie de simulations de dynamique moléculaire pour tester la stabilité du complexe.
Les résultats informatiques, combinés aux données expérimentales antérieures, ont révélé les forces motrices et les mécanismes moléculaires de la liaison des apolipoprotéines aux fibrilles amyloïdes.
Selon les chercheurs, les apolipoprotéines telles que l'apoE sont des « protéines de signature amyloïde » présentes dans les dépôts amyloïdes et influencent probablement leur formation. “Notre travail montre comment ces protéines peuvent interagir directement avec diverses amyloïdes et ainsi favoriser ou bloquer leur croissance et leur prolifération en se liant aux côtés des fibrilles ou à leurs extrémités”, ajoute l'auteur correspondant Olga Gursky, Ph.D., professeur de pharmacologie, physiologie et biophysique. à l'école.
Outre Aβ, près de 40 autres protéines forment des amyloïdes pathologiques dans diverses maladies humaines, notamment la maladie de Parkinson, l'encéphalopathie traumatique chronique, l'amylose à chaînes légères et de nombreux autres troubles potentiellement mortels. Des apolipoprotéines telles que l'apoE se retrouvent dans tous ces dépôts amyloïdes. Ces travaux montrent comment ces protéines peuvent interagir directement avec les amyloïdes et ainsi influencer leurs propriétés physiques et biologiques.
Ces résultats apparaissent en ligne dans Sciences de la vie cellulaire et moléculaire.
Plus d'information:
Emily Lewkowicz et al, La modélisation moléculaire de l'apoE dans des complexes avec des fibrilles amyloïdes-β d'Alzheimer provenant du cerveau humain suggère une base structurelle pour le co-dépôt d'apolipoprotéines avec des amyloïdes, Sciences de la vie cellulaire et moléculaire (2023). DOI : 10.1007/s00018-023-05026-w
Fourni par l'École de médecine de l'Université de Boston
Citation: Les chercheurs déterminent la base structurelle des interactions ApoE-Aβ (7 décembre 2023) récupéré le 7 décembre 2023 sur
Ce document est soumis au droit d'auteur. En dehors de toute utilisation équitable à des fins d'étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni seulement pour information.
